Reacción al estudio que asegura que la gripe H1N1 estacional desciende directamente de la de 1918
Un estudio publicado hoy en la revista Nature Communications analiza la variabilidad genómica del virus de la gripe responsable de la pandemia de 1918 y cómo este dio lugar a la gripe H1N1 estacional posterior.
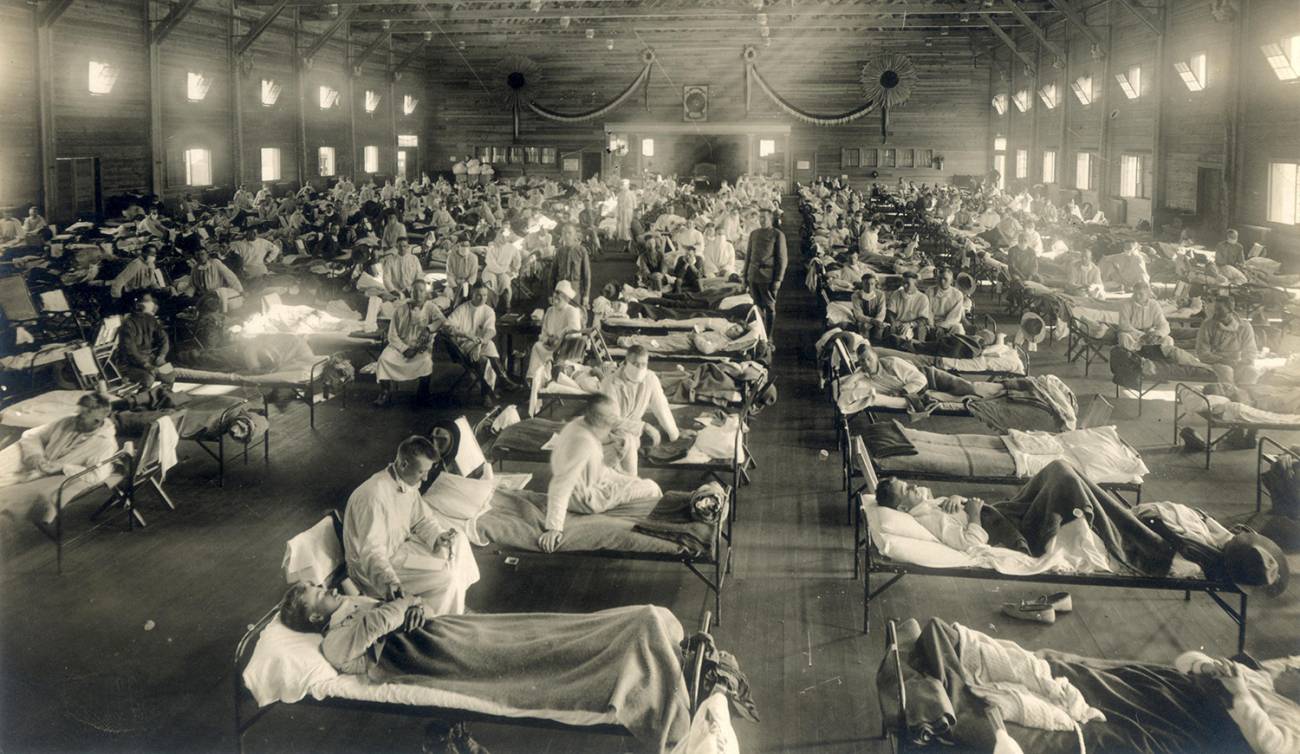
Adrian - 1918
Adrian Hugo Aginagalde
Portavoz de la Sociedad Española de Medicina Preventiva, Salud pública y Gestión sanitaria (SEMPSPGS), jefe de servicio de la Unidad de Vigilancia Epidemiológica e Información Sanitaria de Gipuzkoa y, anteriormente, jefe de servicio de la Unidad de Programas de Cribado Poblacional en el Ministerio de Sanidad
Se trata de un trabajo que intenta, a través de la secuenciación de ARN de muestras de personas fallecidas en aquel momento y la técnica del reloj molecular, determinar el momento de la mutación y el grado de similitud con la gripe H1N1 circulante.
Es compatible con la hipótesis que se ha manejado a nivel histórico hasta el momento. Según esta, debió haber una mutación entre otoño e invierno de 1918 que incrementó su virulencia en comparación con el virus circulante entre primavera y verano de 1918. Se conocía que dicha mutación debía haber ocurrido, aunque no había evidencias de ello, pero que la distancia genética no debía haber sido lo suficientemente alta como para que quienes habían pasado la infección en la primera onda no conservaran la inmunidad en la segunda. Al mismo tiempo, los datos son compatibles con lo que se conoce sobre los virus H1N1 de influenza, es decir, que previamente a 2009 estaban relacionados con el de 1918.
Tiene su interés el que no solo hayan recurrido a muestras de personas enterradas en el permafrost, como ocurría hasta el momento, sino el que hayan incorporado piezas conservadas en los Museos de Historia de la Medicina. Esto permite ampliar el ámbito geográfico a estudio y estudiar mejor las diferencias entre las muestras de virus circulantes a ambos lados del Atlántico.
Confirma que las infecciones respiratorias agudas por virus no desaparecen, sino que desplazan a los serotipos anteriores y se estacionalizan, causando incrementos de la mortalidad en los inviernos siguientes. También que no se alcanza una inmunidad de grupo en estos virus que bloquee la transmisión y que no existe una tendencia natural (o intrínseca) a reducir su virulencia.
Es la desaparición de los factores de riesgo extraordinarios que favorecen su transmisión e incrementan la vulnerabilidad (como una Guerra Mundial), así como la disminución de las personas susceptibles, lo que hace que vaya disminuyendo su impacto; pero sin desaparecer, generando excesos de mortalidad ocasionales mayores de los esperados los inviernos posteriores y sin mutaciones que incrementan su benignidad.
María - 1918
María Iglesias-Caballero
Viróloga, técnica superior especializada en el Laboratorio de Referencia de Virus Respiratorios y Gripe del Centro Nacional de Microbiología, Instituto de Salud Carlos III
La caracterización y estudio del virus de la gripe de la pandemia de 1918 es un campo muy interesante. Nos permite con la metodología actual poder conocer algo más del virus que causó una pandemia que tiene similitudes con la que sufrimos actualmente.
Este trabajo reivindica la importancia de las colecciones archivadas bien conservadas, que son una fuente de material que permite actualizar, con el desarrollo de nuevas metodologías, la información sobre patógenos pasados que pueden aportar información aplicable en el presente y futuro.
La principal limitación del trabajo es el número de muestras empleadas, como es normal dada la dificultad de su obtención. Pese a esta limitación, la metodología empleada tanto para la obtención de las nuevas secuencias del virus por secuenciación masiva como los modelos evolutivos empleados son adecuados y permiten obtener de manera fiable los resultados que exponen.
La comparativa de muestras europeas previas y durante el pico de la pandemia y con respecto a las muestras americanas aporta información sobre la variabilidad viral que podía existir entre los virus pandémicos y las adaptaciones al hospedador que pudieron surgir.
Esta información nos permite a los virólogos de la actualidad vigilar cambios en esas posiciones en virus animales o durante la emergencia de nuevos virus pandémicos. Este trabajo aporta la descripción de cambios que afectan a la actividad de la polimerasa del virus, aunque no sabemos si tiene un impacto real en la replicación de este, y cambios que pueden estar asociados a la adaptación al hospedador en la nucleoproteína y que además mejorarían la capacidad del virus de responder al sistema inmune innato.
En 2014 Rambaut et al. publicó un trabajo que relacionaba el origen del virus con la recombinación de una hemaglutinina de un virus H1 humano circulante junto con 7 segmentos de un virus de la gripe aviar. En este trabajo, que no puede descartar dicha hipótesis, presenta una nueva alternativa. El análisis filogenético, que emplea las nuevas secuencias prepandemia disponibles, revela la asociación robusta de todos los genes internos de los virus estacionales y los pandémicos, excepto en el caso de la hemaglutinina y la neuraminidasa. En el caso de los antígenos virales, estos segmentos se agrupan mejor con los virus porcinos que con los virus de la gripe circulantes hasta el momento. La divergencia evolutiva entre las hemaglutininas y neuraminidasas estacionales y las pandémicas calculada con diferentes modelos evolutivos muestra la posibilidad de un origen diferente al propuesto hasta el momento.
- Artículo de investigación
- Revisado por pares
- Artículo de investigación
- Revisado por pares
