Desarrollan una nueva técnica de edición del genoma que utiliza 'puentes’ de ARN
Dos artículos publicados en Nature describen una nueva técnica de edición del genoma que permite la inserción, inversión o eliminación de secuencias largas de ADN en posiciones del genoma concretas. Se trata de un enfoque en un solo paso que podría proporcionar un método más sencillo de edición genómica en el futuro. Los autores describen una técnica para fabricar recombinasas reprogramables –enzimas clave en la recombinación genética–. Estas enzimas están guiadas por ARN, que actúa como un puente dirigiendo la recombinasa a los sitios diana y facilitando una edición predeterminada.

Montoliu - Puente ARN
Lluís Montoliu
Investigador en el Centro Nacional de Biotecnología (CNB-CSIC) y en el CIBERER-ISCIII
Las herramientas CRISPR-Cas9 de edición genética tienen un potencial extraordinario, pero no son ni infalibles ni totipotentes. Permiten inactivar genes específicos de forma muy eficiente, generando mutaciones; eso es lo que hacen mejor. Pero la sustitución precisa de nucleótidos, de letras del genoma, o la inserción, deleción o inversión de fragmentos de ADN de tamaño considerable lo resuelven mal, con altas dosis de incertidumbre. De ahí que se hayan desarrollado las herramientas CRISPR de segunda generación (los editores de bases) y las de tercera generación (los editores de calidad), presentados por David Liu (Instituto BROAD, Boston) en los años 2016 y 2019, respectivamente. Los editores de bases permiten substituir un nucleótido con robustez y según ciertas combinaciones posibles (A⭢G, C⭢T, C⭢G) y los editores de calidad permiten introducir, eliminar o invertir pequeñas secuencias de ADN. Pero la manipulación de grandes segmentos del genoma, para insertarlos, delecionarlos (escindirlos) o invertirlos sigue siendo un reto todavía, con altos niveles de incertidumbre.
En 2021 el laboratorio de Marc Güell (UPF, Barcelona) presentó una nueva herramienta híbrida, mezclando elegantemente el dominio nucleasa de la proteína Cas9 de un sistema CRISPR con una transposasa (una enzima propia de los transposones, necesaria para que estos elementos móviles puedan escindirse de un sitio y saltar a otro del genoma). A esa nueva herramienta la denominó FiCAT (Find and cut-and-transfer mammalian genome engineering). El sistema FiCAT permitía transferir fragmentos de ADN de tamaño considerable (< 10 kb) con alrededor de un 25-30 % de eficiencia en células de ratón y humanas, aunque dejaba huella del salto, al insertar también las secuencias invertidas y repetidas terminales (ITR) que flanquean al transposón y que este elemento móvil necesita para movilizar el fragmento de ADN a insertar.
El laboratorio de Patrick D. Hsu (Univ. Berkeley, California) ha subido la apuesta con el nuevo sistema de edición que acaba de presentar en sendos artículos publicados en Nature. En el primero de ellos describe esta nueva manera de modificar el ADN mediante una recombinación programable dirigida por una pequeña molécula de ARN, a la que llaman ARN puente (bridge RNA). Hsu describe estos componentes en determinados elementos móviles IS (elementos de inserción de secuencias), en concreto con el elemento móvil IS110, y comprueba que esta pequeña molécula de ARN puente contiene dos secuencias complementarias al ADN de partida (donante) y al ADN de llegada (receptor), que pueden variarse a voluntad para dirigir la inserción de segmentos de ADN de hasta ~5 kb de forma específica a un lugar determinado del genoma. De manera similar, el mismo sistema descrito del elemento móvil IS permite escindir (delecionar) o invertir grandes segmentos de ADN, y esta vez lo hace sin dejar ninguna huella ni cicatriz alguna en el sitio de inserción, de forma limpia, a diferencia del sistema FiCAT. Sorprendente y muy prometedor. Con altas eficiencias de inserción (~85 % eficiencia) aunque también con considerables inserciones en sitios imprevistos del genoma, parecidos, pero no exactos, hasta en un ~30 % de los casos. Obtienen resultados comparables con las deleciones y las inversiones, con un rango de efectividad que varía entre el ~32 % y el ~99 % para las deleciones y entre el ~5 % y el ~98% para las inversiones.
Lo más sorprendente de esta nueva herramienta, que todavía debe optimizarse y mejorarse más, es su alto nivel de compactación, con un ARN puente de apenas 150-250 nucleótidos y una pequeña enzima recombinasa codificada de apenas 300-460 aminoácidos que caben dentro de este elemento móvil IS110. Los resultados presentados solamente se han realizado en bacterias, en Escherichia coli. Ignoramos si estos elementos IS van a funcionar también en células de mamífero, de ratón o humanas. Es previsible que lo hagan, aunque deberíamos esperar a ver estos resultados antes de lanzar las campanas al vuelo demasiado pronto, para un sistema de edición mediante recombinación basado en los elementos móviles IS que promete resolver las carencias de los sistemas CRISPR a la hora de reconstruir las graves alteraciones cromosomales que frecuentemente son causa de enfermedades congénitas.
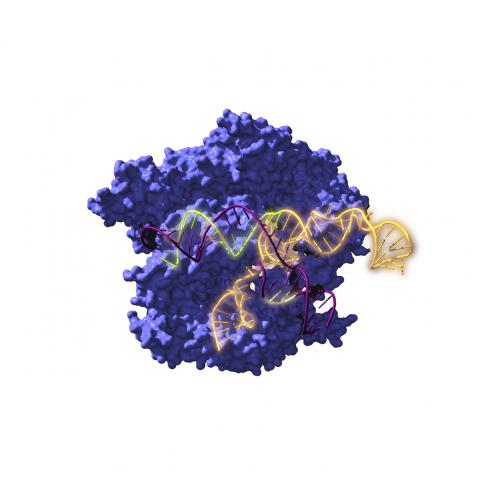
El segundo de los artículos de Hsu describe la ultraestructura de este sistema IS junto con el ARN puente y la diana de ADN y el ADN donante en diferentes momentos de la reacción enzimática. Estos datos permiten reconstruir todos los pasos del proceso, y deducir el mecanismo mediante el cual estos pequeños elementos móviles de la familia IS pueden promover la inserción, deleción o inversión de fragmentos de ADN con elevada eficacia (aunque variable) y de forma limpia, sin dejar rastro, sin arrastrar ni duplicar secuencias colindantes.
Los transposones o elementos genéticos móviles de ADN fueron descubiertos en la década de los años 40 por Barbara McClintock en el genoma del maíz y luego encontrados en prácticamente los genomas de todos los seres vivos. McClintock tuvo que esperar casi 40 años para recibir, en solitario, el merecido premio Nobel de Fisiología o Medicina, en 1983, que reconocía el valor e impacto de su descubrimiento. Las proteínas que median el salto de los transposones son las transposasas, emparentadas con las nucleasas Cas de los sistemas CRISPR y también con estas pequeñas recombinasas de los elementos IS. Creo intuir la sonrisa de Barbara McClintock entre las líneas de los artículos de Hsu, que nos recuerda una vez más que los transposones no solamente han sido y siguen siendo motor del cambio, de la evolución, sino que nos siguen proporcionando nuevos sistemas para cada vez modificar y manipular el genoma de cualquier ser vivo a voluntad con mayor versatilidad y precisión.
- Artículo de investigación
- Revisado por pares
Hiraizumi et al.
- Artículo de investigación
- Revisado por pares
- Artículo de investigación
- Revisado por pares
Durrant et al.
- Artículo de investigación
- Revisado por pares