AlphaGenome, una herramienta de IA de Google, predice el impacto de las variaciones en el ADN
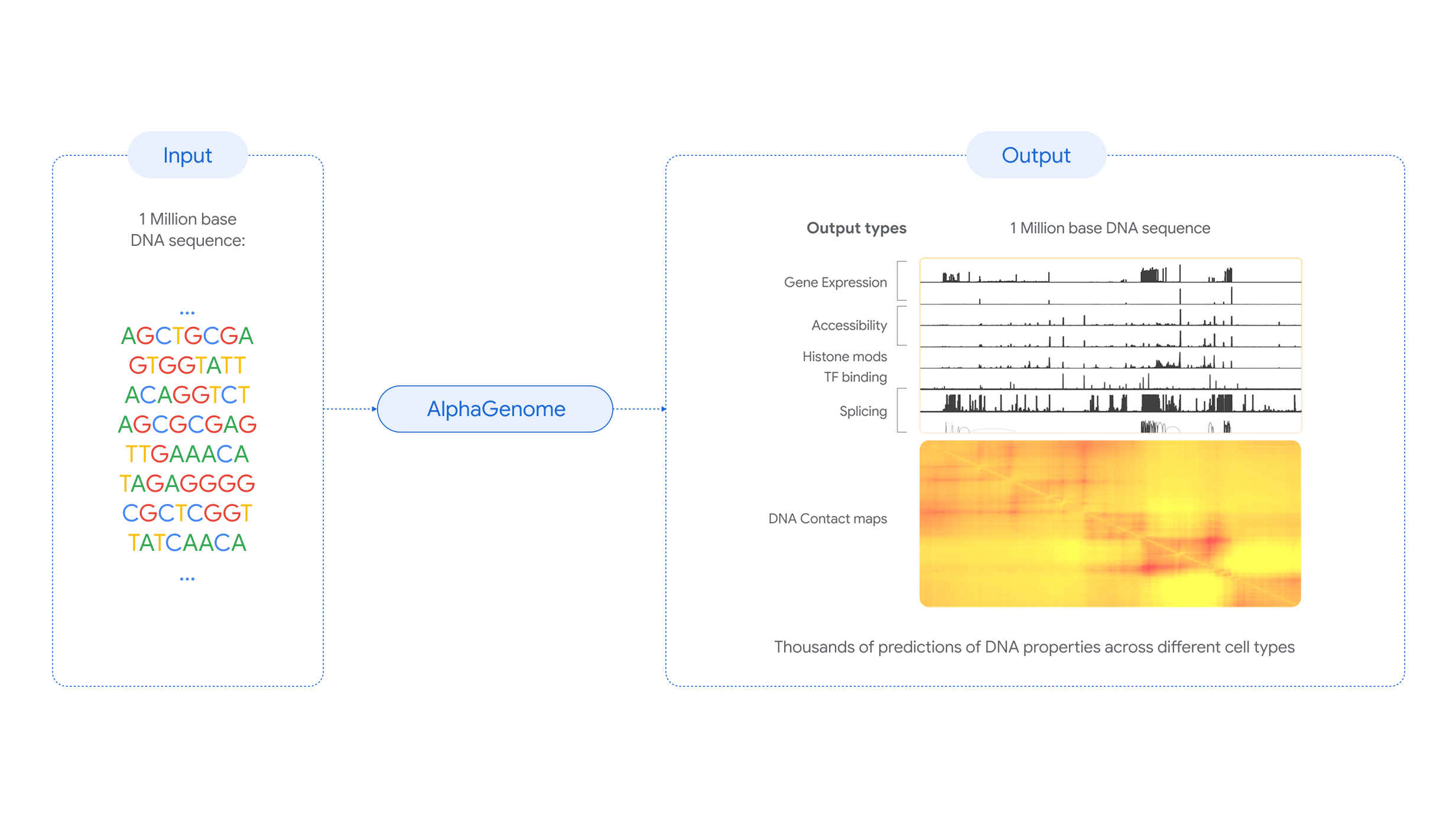
AlphaGenome es un modelo de aprendizaje profundo desarrollado por Google DeepMind capaz de predecir la función de secuencias de ADN de hasta un millón de pares de bases. Una evaluación de la herramienta muestra que iguala o mejora la capacidad de predicción de los modelos existentes en 25 de las 26 pruebas realizadas. Según los autores, que forman parte de la propia Google DeepMind, AlphaGenome puede ayudar a los científicos a “comprender mejor la función del genoma, la biología de las enfermedades y, en última instancia, impulsar nuevos descubrimientos biológicos y el desarrollo de nuevos tratamientos”. Los resultados se publican en Nature.
Montoliu- AlphaGenome
Lluís Montoliu
Investigador en el Centro Nacional de Biotecnología (CNB-CSIC) y en el CIBERER-ISCIII
Un ratón, un rodaballo y una persona tiene el mismo número de genes aproximadamente, alrededor de 20.000 genes cada una de estas especies. Sin embargo, todo el mundo comprende que somos muy diferentes a un ratón y a un rodaballo. Por lo tanto, de alguna manera, el número de genes que tenemos no nos informa de la complejidad o de cómo vamos a ser. La solución a esta paradoja la tenemos que buscar en el genoma no codificante, el que no contiene información que codifica proteínas.
Recordemos que, en todos estos animales, y en particular en nosotros, esos 20.000 genes apenas ocupan un 2 % de todo nuestro genoma. Esto causó durante muchos años una sorpresa a los genetistas. ¿Qué hay en el 98 % restante del genoma? (es decir, ¡en la mayor parte del genoma!). La respuesta es que esa parte oscura y desconocida del genoma alberga muchas secuencias repetitivas, muchos elementos móviles (transposones y retrotransposones) y, lo que es más relevante, las secuencias de ADN reguladoras (a las que se pegan proteínas específicas) que le indican a un gen cuándo y dónde debe empezar a funcionar, o cuándo y dónde debe apagarse. En otras palabras, la complejidad y diversidad morfológica que tenemos un ratón, un rodaballo y una persona no se consigue aumentando o disminuyendo el número de genes, sino alterando precisamente esas secuencias reguladoras, las que harán que los mismos genes se activen o desactiven en momentos distintos del desarrollo o en células diferentes, según la especie. Esencialmente, si cambiamos el programa de activación y desactivación con los mismos genes generaremos animales muy diferentes. Y eso es lo que finalmente produce animales tan dispares como esos tres, a pesar de que tengan un número de genes similar.
Durante años se han desarrollado algoritmos y programas informáticos que analizaban esta parte del genoma no codificante, sin sentido, a la búsqueda de secuencias precisas que supiéramos, por otras investigaciones, que son elementos reguladores. Los hay de muchos tipos, los hay que están asociados a fenómenos de activación génica, otros a fenómenos de silenciamiento o hasta algunos se comportan como aisladores, y separan lo que ocurre en un gen de lo que le vaya a pasar al gen vecino. Estos programas predictivos se basan en la comparación sistemática de secuencias que sabemos operan como activadores, silenciadores o aisladores (entre otros muchos tipos de elementos reguladores) en una determinada secuencia problema de ADN, que normalmente no puede ser demasiado larga, de unos pocos miles de nucleótidos. Normalmente cada uno de esos programas se especializa en la detección de cada uno de estos tipos de elementos reguladores.
Todo lo anterior ha saltado por los aires con la aparición de AlphaGenome, cuya presentación en sociedad acaba de publicar la revista Nature. Una nueva inteligencia artificial de Google, otra más, que es capaz de realizar todas esas inspecciones y deducciones sobre prácticamente todos los tipos de elementos reguladores, todos a la vez, y es capaz de hacerlo sobre enormes segmentos de ADN, de hasta un millón de letras, algo que no sabíamos hacer.
Google ya sorprendió a la profesión científica hace unos pocos años con AlphaFold y su impresionante capacidad de predecir la estructura y plegamiento de las proteínas a partir solamente de la secuencia de ADN. Ahora nos vuelve a dejar boquiabiertos con AlphaGenome y su capacidad de interpretar y predecir todas aquellas secuencias del genoma sin sentido, del genoma no codificante, que se hallan en un segmento enorme de nuestro genoma. Para desarrollar esta IA los investigadores la han entrenado analizando el genoma humano y el del ratón.
Naturalmente esto tendrá un impacto significativo no solamente en investigación básica, para entender cómo funcionan los genes, sino también en aspectos más prácticos, aplicados. Por ejemplo, cómo identificar nuevas secuencias de ADN que sean relevantes en esas zonas del genoma que habitualmente se desdeñan o no se tenían en cuenta. La información que proporcionará AlphaGenome habrá que tenerla muy en cuenta para analizarla cuidadosamente cuando abordemos los diagnósticos genéticos.
Una alteración en alguno de estos elementos reguladores, que impida la activación o el silenciamiento de un gen cuando debería ocurrir, puede tener como consecuencia un cambio en el patrón de desarrollo embrionario o la aparición de síntomas de alguna patología asociada precisamente al funcionamiento anómalo de ese gen (sin que la secuencia del propio gen haya cambiado en absoluto). En este caso, la mutación no estaría dentro del gen sino fuera, en secuencias más o menos alejadas, en las secuencias de ADN que ocultan estos elementos reguladores y que ahora esta IA llamada AlphaGenome descubrirá eficazmente en cualquier segmento del genoma que queramos analizar, especialmente, en el genoma no codificante, el genoma que llamábamos sin sentido (cuando no lo entendíamos) y que ahora, gracias a AlphaGenome, podremos empezar a descifrar con mucha mayor precisión y detalle del que hasta ahora conocíamos.
Goldstone - AlphaGenome
Robert Goldstone
Director de Genómica en el Francis Crick Institute (Londres, Reino Unido)
AlphaGenome de DeepMind representa un hito importante en el campo de la inteligencia artificial (IA) genómica. Este nivel de resolución, en particular para el ADN no codificante, supone un avance que lleva la tecnología del interés teórico a la utilidad práctica, permitiendo a los científicos estudiar y simular programáticamente las raíces genéticas de enfermedades complejas.
El modelo funciona excepcionalmente bien en tareas que podrían regirse por reglas ‘gramaticales’ rígidas escritas en el ADN, como la predicción del sitio de empalme. En estas áreas está listo para reemplazar de inmediato las herramientas estándar más antiguas. Y una de las demostraciones más notables es su capacidad para predecir la expresión génica únicamente a partir de la secuencia de ADN. Si bien no es perfecto, dado que la expresión génica se ve influida por factores ambientales complejos que el modelo no puede detectar, lograr el nivel de precisión demostrado, basado únicamente en reglas ‘locales’ del ADN, es una hazaña técnica increíble.
AlphaGenome no es una solución mágica para todas las cuestiones biológicas, pero sí una herramienta fundamental y de alta calidad que convierte el código estático del genoma en un lenguaje descifrable para el descubrimiento.
Conflictos de interés: no hay declaraciones de conflictos de interés. Hay un laboratorio de DeepMind en el Instituto Francis Crick, pero Robert Goldstone no ha trabajado en AlphaGenome.
Lehner - AlphaGenome
Ben Lehner
Director de Genómica Generativa y Sintética del Instituto Wellcome Sanger de Cambridge (Reino Unido)
AlphaGenome es un excelente ejemplo de cómo la IA está acelerando el descubrimiento biológico y el desarrollo de terapias. Identificar las diferencias precisas en nuestros genomas que nos hacen más o menos propensos a desarrollar miles de enfermedades es un paso clave para desarrollar mejores terapias. AlphaGenome y modelos similares, que ayudan a descifrar el código regulador de nuestro genoma, facilitarán enormemente este proceso.
Como ya esperábamos de Google Deepmind, AlphaGenome es una gran obra de ingeniería que reúne ideas desarrolladas por diversos científicos en un modelo que marca la pauta. En el Instituto Wellcome Sanger hemos probado AlphaGenome con más de medio millón de nuevos experimentos y, sin duda, funciona muy bien. Sin embargo, AlphaGenome dista mucho de ser perfecto y aún queda mucho trabajo por hacer. La calidad de los modelos de IA depende de los datos utilizados para entrenarlos. La mayoría de los datos existentes en biología no son muy adecuados para la IA: los conjuntos de datos son demasiado pequeños y no están bien estandarizados. El mayor desafío ahora mismo es cómo generar los datos para entrenar la próxima generación de modelos de IA aún más potentes. Necesitamos hacerlo de forma rápida, rentable y de forma que tanto los datos como los modelos resultantes estén disponibles para todos.
Conflictos de interés: Ben Lehner recibe financiación para investigación de Google DeepMind a través de una pequeña colaboración con ellos. No es autor ni participó en el desarrollo del modelo AlphaGenome.
- Artículo de investigación
- Revisado por pares
Avsec et al.
- Artículo de investigación
- Revisado por pares